从原理上来讲,虚拟筛选可以分为两类,即基于受体(Receptor-based virtual screening)的虚拟筛选和基于配体(ligand-based virtual screening)的虚拟筛选。
基于受体的虚拟筛选也称为基于结构的虚拟筛选,利用分子对接技术,基于受体的三维结构,在结合位点处自动的匹配化合物数据库中的小分子,然后对可能的结合模式运用基于分子力场的打分函数进行结合能计算,最终得到化合物能量排名。
前两期教程中为大家分享了基于结构的虚拟筛选步骤中的文件导入、生成结合位点以及分子对接的操作,本次教程以上期教程的对接结果为例进行结果的查看与分析以及结合位点的表征。
①对接结果可视化
在软件界面右上角点击Tasks > Browse > Receptor-Based Virtual Screening > Pose Viewer打开对接姿势面板;
点击Import File选择导入并打开上期对接产生的glide_1FJS_screening_pv.maegz文件;点击Set Up;
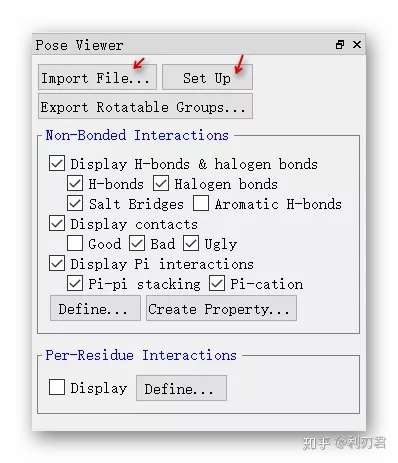
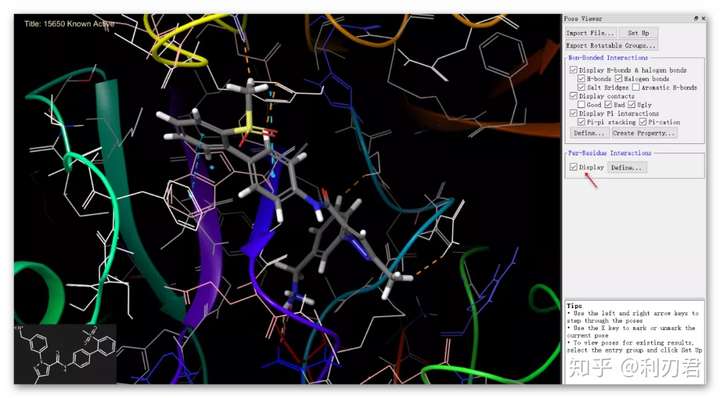
在Per Residue Interactions选项下方, 勾选Display;此时残基根据它们的相互作用能被着色,从红色(不利)到蓝色(有利)。
- 注:可通过键盘的←和→来浏览不同的配体
②对接结果分析
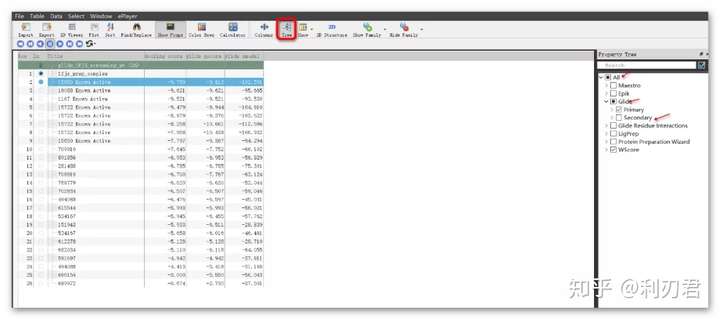
在项目表Project Table中,单击属性树Tree图标,属性树出现在项目表的右侧;
点击All框两次,此时所有方框均不选;
点击Glide,点击Secondary,此时在项目表中就只显示了Glide的基本性质;
可在表中看到对接结果的docking score、glide gscore以及glide emodel的详细数值。
③pre-docked XP结果可视化
在软件界面右上角点击Tasks > Browse > Receptor-Based Virtual Screening > Visualize XP Interactions打开XP Visualizer窗口;点击Open;
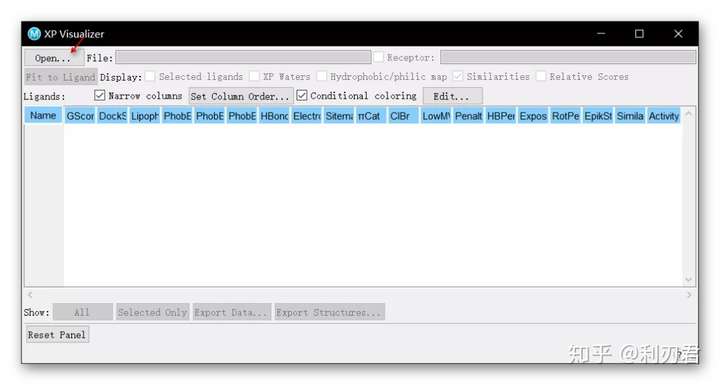
选择factorXa_xp_refine_pv.maegz文件,点击Open打开;
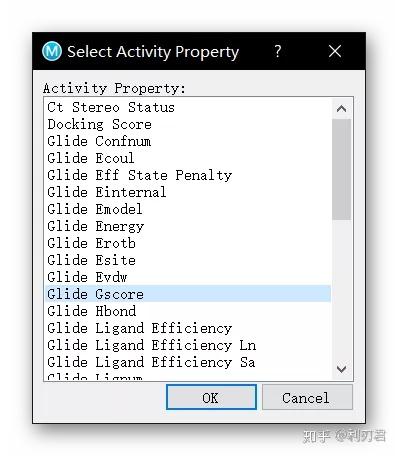
在弹出的Select Activity Property对话框中选择Glide Gscore作为活动属性, 点击OK;
得分函数的个别项目被标记为红色(惩罚)或蓝色(奖励),以此我们可分析对接结果评价中的各相互作用的贡献情况。
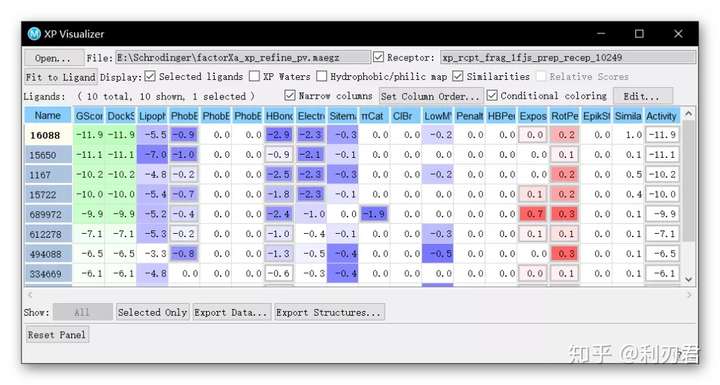
点击Export Data可将该表格导出为.csv格式文件;
鼠标单击表格中的彩色条目可以使奖励情况在工作区中显示;
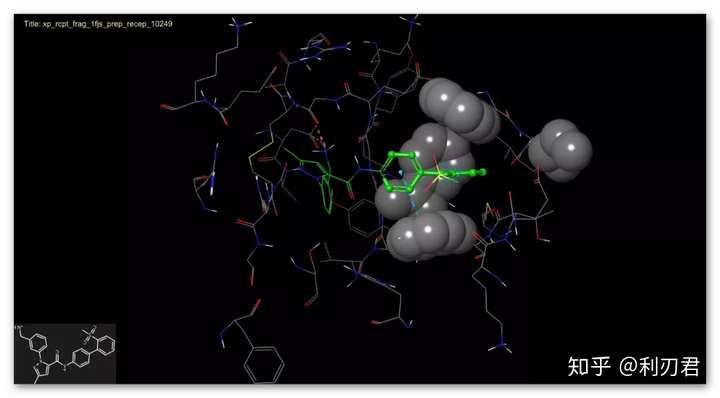
④使用SiteMap分析结合位点
双击项目列表中的1fjs_prep_complex前面的小圈以固定显示;
在软件右上角点击Tasks > Browse > Structure Analysis > Binding Site Detection打开SiteMap工具;
在Task下方,勾选Evaluate a Single binding site region;
在工作界面中点击配体分子以选中;更改任务名称Job name为:sitemap_1fjs;
点击Run运行任务;
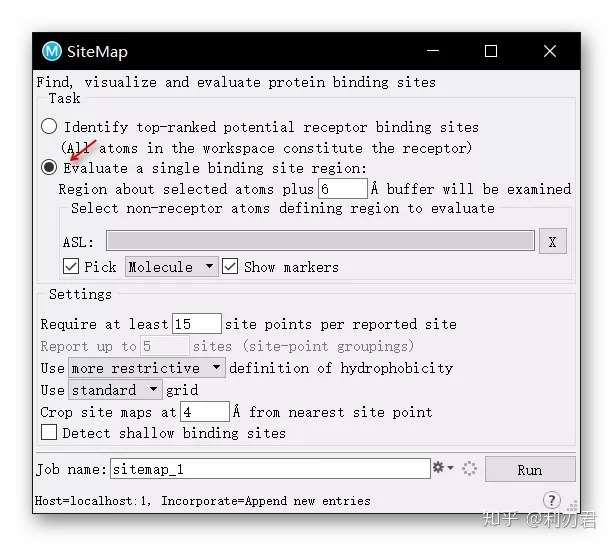
任务完成后,新的条目会出现在项目列表中,勾选sitemap_1fjs_site_1以查看位点1;
此时,工作界面出现不同的表面,表示不同的亲水性区域:疏水性(黄色),受体(红色),供体(蓝色)。
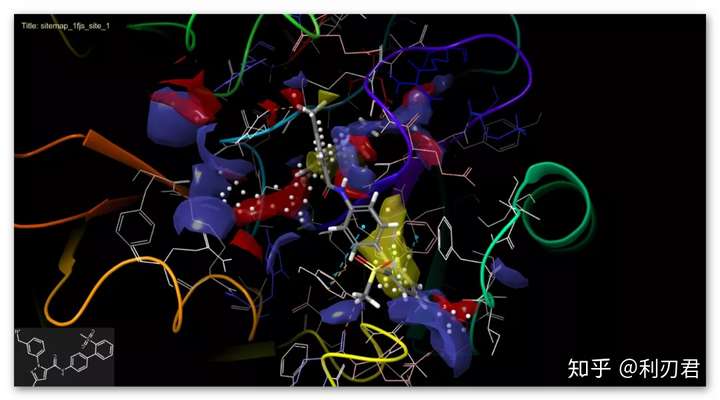
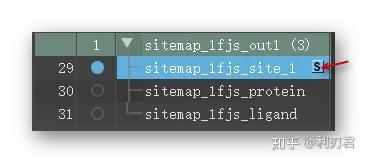
在条目列表中,单击sitemap_1fjs_site1旁边的S,可切换与位点地图关联的表面。
以上就是利用借助Schrodinger软件进行基于结构的虚拟筛选的教程下篇~







