从原理上来讲,虚拟筛选可以分为两类,即基于受体(Receptor-based virtual screening)的虚拟筛选和基于配体(ligand-based virtual screening)的虚拟筛选。
基于受体的虚拟筛选也称为基于结构的虚拟筛选,利用分子对接技术,基于受体的三维结构,在结合位点处自动的匹配化合物数据库中的小分子,然后对可能的结合模式运用基于分子力场的打分函数进行结合能计算,最终得到化合物能量排名。
上期教程为大家分享了基于结构的虚拟筛选步骤中的文件导入及生成结合位点的操作,本次教程以软件的实例进行基于分子对接方法的虚拟筛选的步骤演示。
①原配体分子准备
在进行虚拟筛选之前,我们往往需要先对复合物中的原配体进行对接,来考察对接方法的可靠性。
上期教程中打开的文件中包含名称为1fjs_prep_lig的配体分子,该分子为受体蛋白的原配体分子;
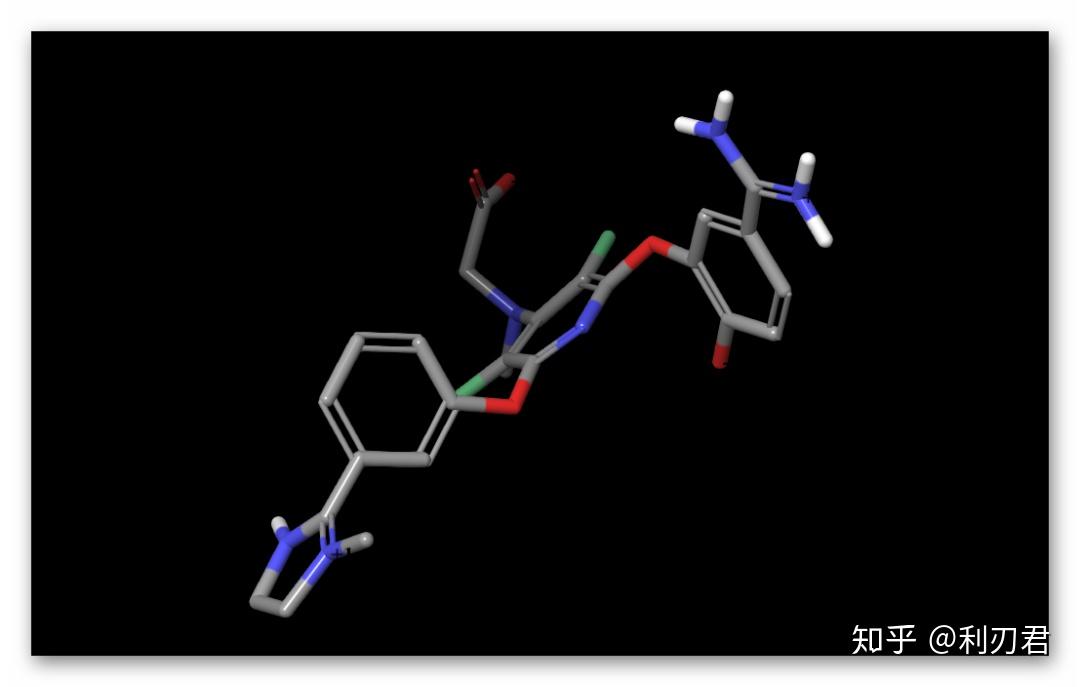
在软件右上角点击Tasks > Browse > LigPrep,打开配体准备窗口;
在Use structures from选项中, 选择Workspace (1 included entry);在Stereoisomers下方的选项中, 勾选Determine chiralities from 3D structure;
更改任务名称为ligprep_1FJS;
点击Run运行任务,即完成配体的准备工作。
②对接同源配体
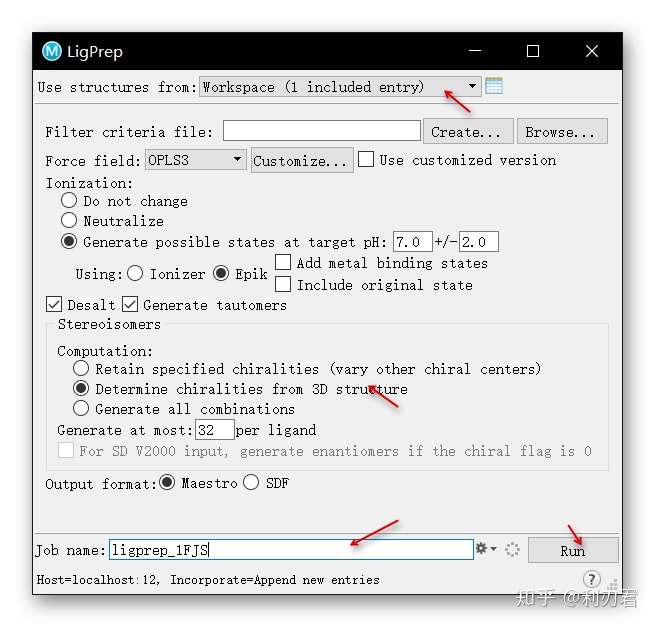
在软件界面右上角点击Tasks > Browse > Receptor-Based Virtual Screening > Ligand Docking,打开分子对接参数设置对话框;
在Receptor grid选项, 点击Browse打开并选择上期活性位点定义产生的glide-grid_1fjs.zip文件;
在Ligands选项卡中, Use ligands from, 下拉菜单选择为Files;
在File name选项中, 点击Browse打开并选择上步产生的准备好的配体文件ligprep_1FJS-out.maegz;
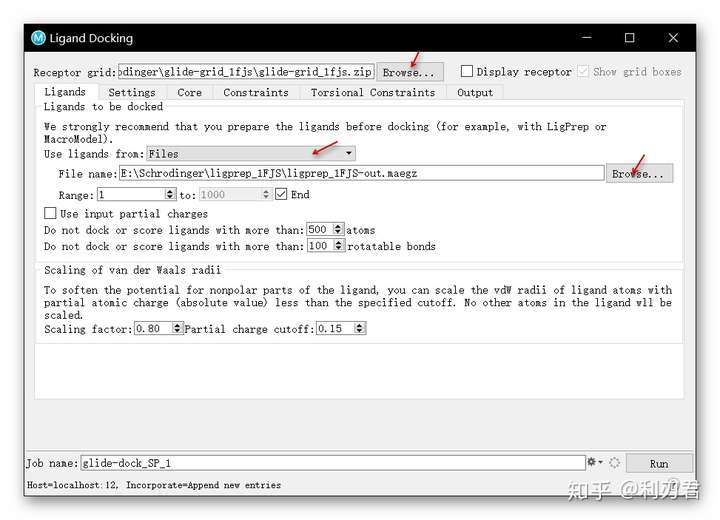
点击Constraints选项卡,在Use下方, 勾选为氨基酸残基ASP 189设定好的氢键约束;
更改任务名称为glide_1FJS_cognate;
点击Run运行任务,任务完成后,项目列表中会出现对接结果文件。
双击1fjs_prep_complex前面的圆圈来固定复合物显示在工作区域;
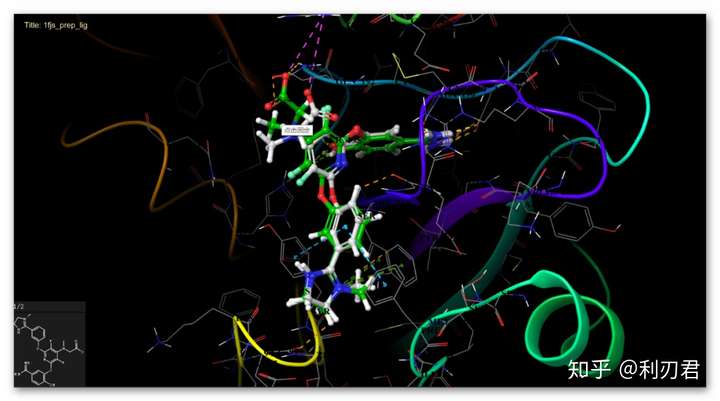
在对接结果glide_1FJS_cognate-pv1组中,一共产生了3个不同的分子构象,可以依次浏览各构象的对接结果;

③对接需筛选的分子
鼠标点击选中50ligs_epik组,该组包括50个小分子,已经经过Ligpre处理,得到91个分子构象,下面我们通过分子对接的方法对这91个分子进行虚拟筛选;
在Ligand Docking对话框中, 点击Ligands选项卡;
在Receptor grid选项, 点击Browse打开并选择上期活性位点定义产生的glide-grid_1fjs.zip文件;
在ligands from选项中, 选择Project Table (selected entries),即选中的91个分子;
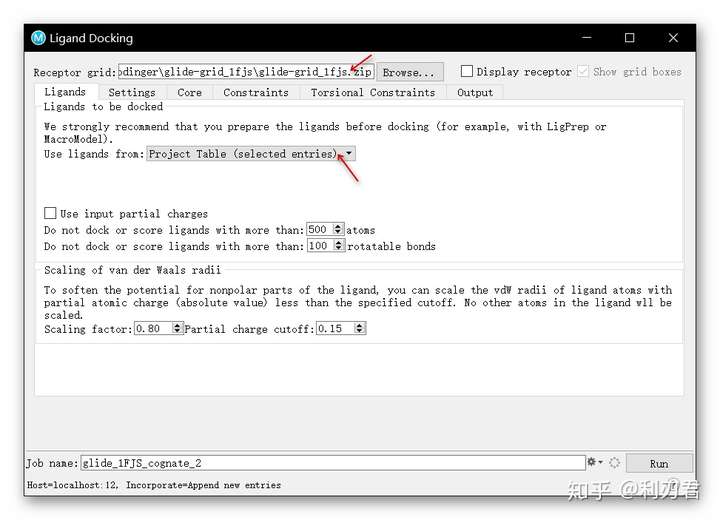
点击Output选项卡,勾选Write per-residue interaction scores;
更改任务名称为glide_1FJS_screening;
点击Run运行任务,等待任务完成即可。

以上就是利用借助Schrodinger软件进行基于结构的虚拟筛选的教程中篇,下篇教程将为大家分享虚拟筛选的结果分析教程~







